
機械学習向けのノートブックをそのままパイプラインに組み込めるSageMaker Notebook JobをPython SDKから使ってみた
この記事は公開されてから1年以上経過しています。情報が古い可能性がありますので、ご注意ください。
こんちには。
データ事業本部 インテグレーション部 機械学習チームの中村( @nokomoro3 )です。
今回は、データサイエンティストや研究者などが作成したノートブックをそのまま機械学習パイプラインに組み込める、SageMaker Notebook Jobについてご紹介します。
仕様の確認
Notebook Jobの使用方法
以下を見る限り、
Notebook Jobは以下の2つの使い方があります。
- Studio上のUIでJupyterLabからジョブを作成する
- SageMaker Python SDKでSageMaker Pipelines経由でオンデマンドもしくはスケジューリング実行する
1点目については、弊社のブログでも記載されています。
今回は自動化などを想定したいので、2点目のSDK経由で実施します。SDK経由でNotebook Jobを使うには、SageMaker Pipelinesを使う必要があります。
ドキュメントの実行例
ドキュメントでは以下のように記載されています。
notebook_job_step = NotebookJobStep(
input_notebook="input-notebook",
image_uri="image-uri",
kernel_name="kernel-name"
)
input_notebook はノートブックのパスを指定すると、S3経由でProcessing Jobとしてアップロードされます。
image_uri はNotebook Jobで使用するコンテナイメージを指定します。以下から情報を探してみてください。
今回は標準的な構成を試すため、sagemaker-distribution-prodのものを使用します。
885854791233.dkr.ecr.us-east-1.amazonaws.com/sagemaker-distribution-prod:2.1-gpu
kernel_name はJupyter上のカーネル名のことです。今回は調べる方法が分からなかったので、JupyterLab Spaceとして sagemaker-distribution イメージでスペースを起動し、シェルで以下を入力することで確認しました。
jupyter kernelspec list
# Available kernels:
# glue_pyspark /opt/conda/share/jupyter/kernels/glue_pyspark
# glue_spark /opt/conda/share/jupyter/kernels/glue_spark
# pysparkkernel /opt/conda/share/jupyter/kernels/pysparkkernel
# python3 /opt/conda/share/jupyter/kernels/python3
# sparkkernel /opt/conda/share/jupyter/kernels/sparkkernel
今回は python3 を設定すれば良さそうです。
パラメータをどのように渡すか
パラメータとして何かしらの情報を渡すために使えそうな引数が無いか以下を確認してみます。
この中では parameters と environment_variables が使用できそうですが、 parameters は裏で実行されるTrainingJobの hyperParameters に設定され、 environment_variables は environment に設定されます。
(以下の裏で実行されるTrainingJobのCloudTrailの例を示します)
{
"requestParameters": {
"hyperParameters": {
// ...
},
"environment": {
// ...
},
}
}
environment に設定されたものは、ノートブック上でも環境変数としてアクセスすることができます。また hyperParameters はノートブック上に変数として埋め込まれるようですのでこちらを活用することも可能です。
今回の記事では、学習したモデルの出力先のS3パスなどを environment_variables に設定してみます。
やってみた
実行環境
Studio上のJupyterLab Spaceやローカル環境からも実施できます。各モジュールのバージョンは以下です。
pydantic 2.9.0
pydantic-core 2.23.2
sagemaker 2.230.0
sagemaker-core 1.0.3
scikit-learn 1.5.1
ジョブ用のノートブック作成
ジョブ用のノートブックを以下に沿って作成しておきます。
まずはPyTorchのサンプルモデルの作成です。
### サンプルモデル作成
import torch
import torch.nn as nn
import torch.optim as optim
import torchvision
import torchvision.transforms as transforms
from torch.utils.data import DataLoader
# GPUが利用可能か確認
device = torch.device('cuda' if torch.cuda.is_available() else 'cpu')
print(f'Using device: {device}')
# データの前処理
transform = transforms.Compose([
transforms.ToTensor(),
transforms.Normalize((0.1307,), (0.3081,))
])
# データセットのロード
train_dataset = torchvision.datasets.MNIST(root='./data',
train=True,
transform=transform,
download=True)
test_dataset = torchvision.datasets.MNIST(root='./data',
train=False,
transform=transform)
# データローダーの設定
train_loader = DataLoader(train_dataset, batch_size=64, shuffle=True)
test_loader = DataLoader(test_dataset, batch_size=1000, shuffle=False)
# モデルの定義
class ConvNet(nn.Module):
def __init__(self):
super(ConvNet, self).__init__()
self.conv1 = nn.Conv2d(1, 32, 3)
self.pool = nn.MaxPool2d(2, 2)
self.conv2 = nn.Conv2d(32, 64, 3)
self.fc1 = nn.Linear(64 * 5 * 5, 128)
self.fc2 = nn.Linear(128, 10)
self.relu = nn.ReLU()
def forward(self, x):
x = self.pool(self.relu(self.conv1(x)))
x = self.pool(self.relu(self.conv2(x)))
x = x.view(-1, 64 * 5 * 5)
x = self.relu(self.fc1(x))
x = self.fc2(x)
return x
# モデルのインスタンス化とGPUへの転送
model = ConvNet().to(device)
criterion = nn.CrossEntropyLoss()
optimizer = optim.Adam(model.parameters(), lr=0.001)
# 学習ループ
num_epochs = 5
for epoch in range(num_epochs):
model.train()
running_loss = 0.0
for i, (images, labels) in enumerate(train_loader):
images = images.to(device)
labels = labels.to(device)
# Forward pass
outputs = model(images)
loss = criterion(outputs, labels)
# Backward pass and optimize
optimizer.zero_grad()
loss.backward()
optimizer.step()
running_loss += loss.item()
if (i + 1) % 100 == 0:
print(f'Epoch [{epoch+1}/{num_epochs}], Step [{i+1}/{len(train_loader)}], Loss: {running_loss/100:.4f}')
running_loss = 0.0
# テストデータでの評価
model.eval()
with torch.no_grad():
correct = 0
total = 0
for images, labels in test_loader:
images = images.to(device)
labels = labels.to(device)
outputs = model(images)
_, predicted = torch.max(outputs.data, 1)
total += labels.size(0)
correct += (predicted == labels).sum().item()
print(f'Accuracy on test set: {100 * correct / total:.2f}%')
print('Finished Training')
# モデルの保存
torch.save(model.state_dict(), 'mnist_cnn.pth')
学習したモデルをSageMakerの他の処理で使用するため、S3にアップロードする処理も追加しておきます。
### S3へのモデルアップロード
import sagemaker
import boto3
import os
# SageMaker セッションの初期化
session = sagemaker.Session()
role = sagemaker.get_execution_role()
region = session.boto_region_name
# モデルの保存パス
model_path = 'mnist_cnn.pth'
# モデルファイルをtar.gzに圧縮
os.system(f'tar -czvf model.tar.gz {model_path}')
s3_uri = os.getenv("MODEL_OUTPUT_S3_URI")
bucket, key_prefix, path = s3_uri[5:].split("/")[0], "/".join(s3_uri[5:].split("/")[1:-1]), s3_uri[5:].split("/")[-1]
# S3にアップロード
session.upload_data(
path=path,
bucket=bucket,
key_prefix=key_prefix
)
アップロード先の MODEL_OUTPUT_S3_URI はジョブの環境変数で渡すようにします。
パイプラインの作成と実行
まずは関連する変数を定義します。
roleはStudio内の環境を使うか、ローカル環境から実施するかで異なるので必要に応じて変更ください。
import sys
import boto3
import sagemaker
from sagemaker.workflow.pipeline_context import PipelineSession
sagemaker_session = sagemaker.session.Session()
region = sagemaker_session.boto_region_name
# role = sagemaker.get_execution_role() # Studio内でやる場合はこちら
role = "{SageMakerの実行ロール}"
pipeline_session = PipelineSession()
default_bucket = sagemaker_session.default_bucket()
次にNotebookJobをPipeline化します。ここで MODEL_OUTPUT_S3_URI を渡します。
from sagemaker.workflow.notebook_job_step import NotebookJobStep
notebook_job_step = NotebookJobStep(
input_notebook="./job.ipynb",
image_uri="885854791233.dkr.ecr.us-east-1.amazonaws.com/sagemaker-distribution-prod:2.1-gpu",
kernel_name="python3",
instance_type="ml.g4dn.2xlarge",
role=role,
environment_variables={
"MODEL_OUTPUT_S3_URI": f"s3://{default_bucket}/sample-notebook-job/models/mnist_cnn/model.tar.gz",
},
)
最後にpipelineを作成し、実行します。
from sagemaker.workflow.pipeline import Pipeline
pipeline_name = f"sample-notebook-job-pipeline"
# 作成
pipeline = Pipeline(
name=pipeline_name,
steps=[notebook_job_step],
sagemaker_session=sagemaker_session,
)
# 登録
pipeline.upsert(role_arn=role)
# 実行
execution = pipeline.start()
# 完了待ち
execution.wait()
実行結果の確認
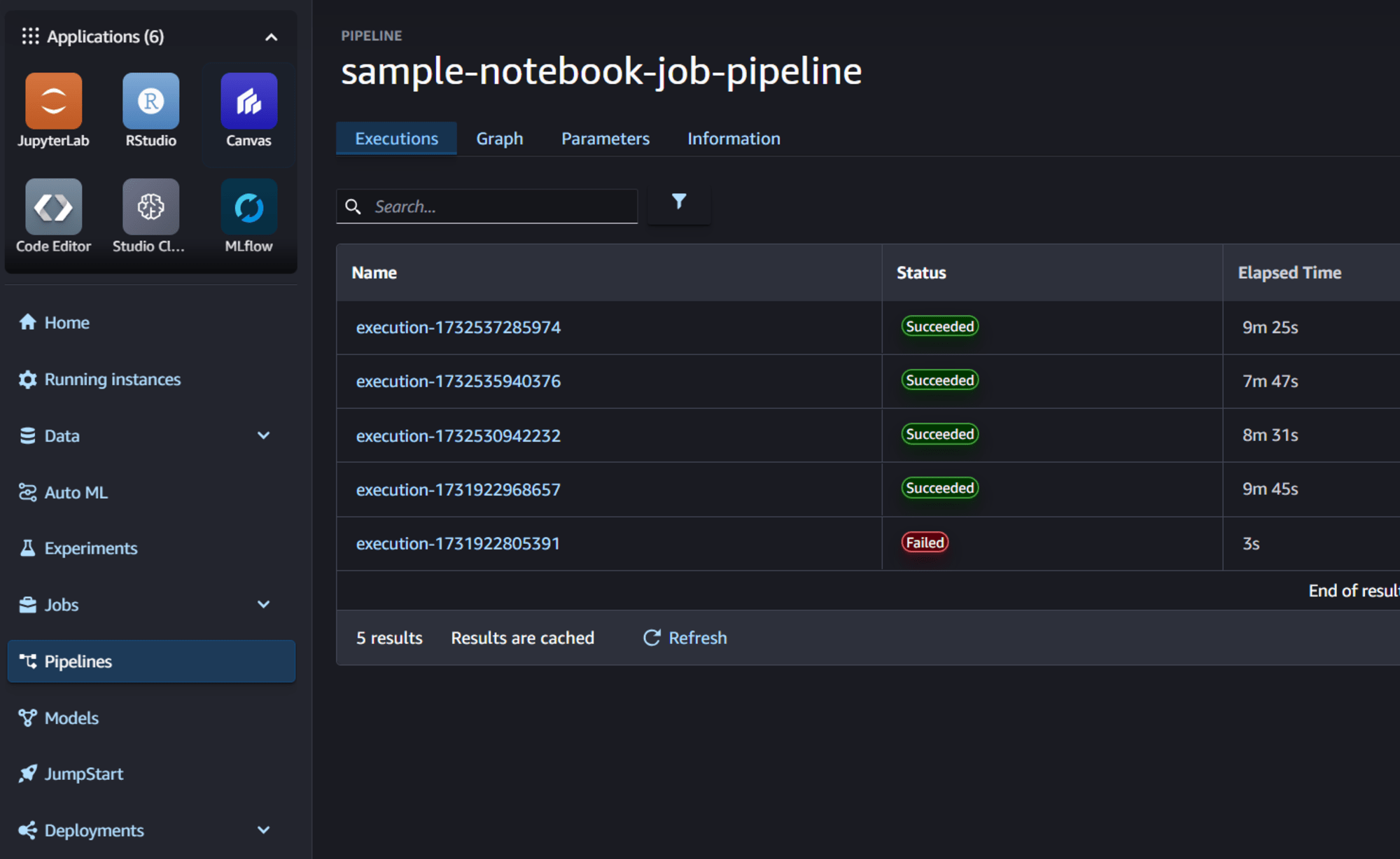
pipelineの結果確認
SageMaker Studioの画面に移動すると、pipelineの実行結果を確認することができます。
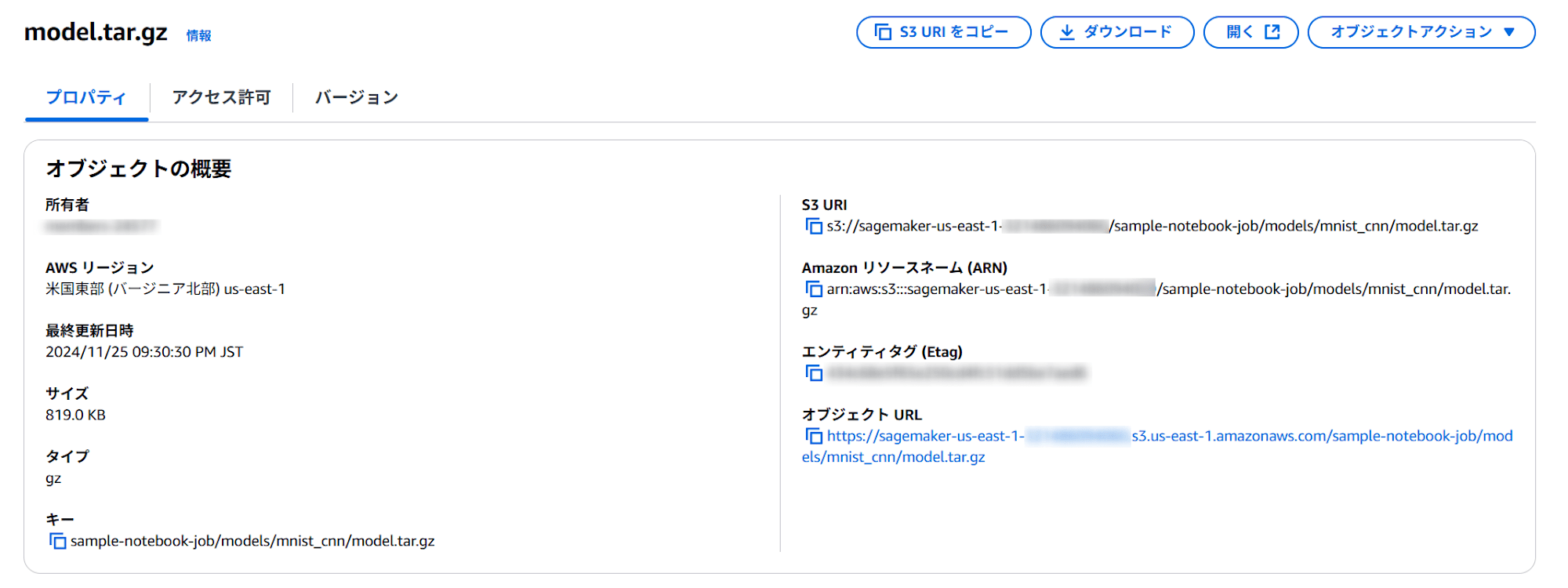
モデルの出力
モデルが以下に出力されていることを確認できました。
s3://{デフォルトバケット}/sample-notebook-job/models/mnist_cnn/model.tar.gz
CloudTrail
裏側で実行されるTraningJobのCloudTrailでのイベントは以下のように記録されていました。
{
"eventVersion": "1.10",
"eventTime": "2024-11-25T12:21:27Z",
"eventSource": "sagemaker.amazonaws.com",
"eventName": "CreateTrainingJob",
"awsRegion": "us-east-1",
"sourceIPAddress": "pipelines.sagemaker.amazonaws.com",
"userAgent": "pipelines.sagemaker.amazonaws.com",
"requestParameters": {
"trainingJobName": "job-ipynb-2024-11-25-12-21-20-016-s5gm5lwv86wz-EaMV5Wq3YO",
"algorithmSpecification": {
"trainingImage": "885854791233.dkr.ecr.us-east-1.amazonaws.com/sagemaker-distribution-prod:2.1-gpu",
"trainingInputMode": "File",
"enableSageMakerMetricsTimeSeries": false,
"containerEntrypoint": [
"amazon_sagemaker_scheduler"
]
},
"roleArn": "{指定した実行ロール}",
"inputDataConfig": [
{
"channelName": "sagemaker_headless_execution_pipelinestep",
"dataSource": {
"s3DataSource": {
"s3DataType": "S3Prefix",
"s3Uri": "s3://{デフォルトバケット}/sample-notebook-job-pipeline/job-ipynb-2024-11-25-12-21-20-016/input-2024-11-25-12-21-24-290",
"s3DataDistributionType": "FullyReplicated"
}
},
"enableFFM": false
}
],
"outputDataConfig": {
"s3OutputPath": "s3://{デフォルトバケット}/sample-notebook-job-pipeline/s5gm5lwv86wz/job-ipynb-2024-11-25-12-21-20-016",
"removeJobNameFromS3OutputPath": false,
"disableModelUpload": false
},
"resourceConfig": {
"instanceType": "ml.g4dn.2xlarge",
"instanceCount": 1,
"volumeSizeInGB": 30
},
"stoppingCondition": {
"maxRuntimeInSeconds": 172800
},
"tags": [
{
"key": "sagemaker:is-studio-archived",
"value": "false"
},
{
"key": "sagemaker:name",
"value": "job-ipynb-2024-11-25-12-21-20-016"
},
{
"key": "sagemaker:pipeline-step-name",
"value": "job-ipynb-2024-11-25-12-21-20-016"
},
{
"key": "sagemaker:pipeline-execution-arn",
"value": "arn:aws:sagemaker:us-east-1:{アカウントID}:pipeline/sample-notebook-job-pipeline/execution/s5gm5lwv86wz"
},
{
"key": "sagemaker:notebook-name",
"value": "job.ipynb"
},
{
"key": "sagemaker:notebook-job-origin",
"value": "PIPELINE_STEP"
}
],
"enableNetworkIsolation": false,
"enableInterContainerTrafficEncryption": true,
"enableManagedSpotTraining": false,
"experimentConfig": {
"experimentName": "sample-notebook-job-pipeline",
"trialName": "s5gm5lwv86wz"
},
"disableEFA": false,
"environment": {
"MODEL_OUTPUT_S3_URI": "s3://sagemaker-us-east-1-{アカウントID}/sample-notebook-job/models/mnist_cnn/model.tar.gz",
"SM_EXECUTION_INPUT_PATH": "/opt/ml/input/data/sagemaker_headless_execution_pipelinestep",
"SM_INPUT_NOTEBOOK_NAME": "job.ipynb",
"SM_KERNEL_NAME": "python3",
"SM_OUTPUT_NOTEBOOK_NAME": "job-ipynb-2024-11-25-12-21-20-016.ipynb",
"AWS_DEFAULT_REGION": "us-east-1",
"SM_SKIP_EFS_SIMULATION": "true",
"SM_ENV_NAME": "sagemaker-default-env",
"SM_JOB_DEF_VERSION": "1.0"
},
"retryStrategy": {
"maximumRetryAttempts": 1
},
"trainingJobArn": "arn:aws:sagemaker:us-east-1:{アカウントID}:training-job/job-ipynb-2024-11-25-12-21-20-016-s5gm5lwv86wz-EaMV5Wq3YO",
"withWarmPoolValidationError": false
},
"responseElements": {
"trainingJobArn": "arn:aws:sagemaker:us-east-1:{アカウントID}:training-job/job-ipynb-2024-11-25-12-21-20-016-s5gm5lwv86wz-EaMV5Wq3YO"
}
}
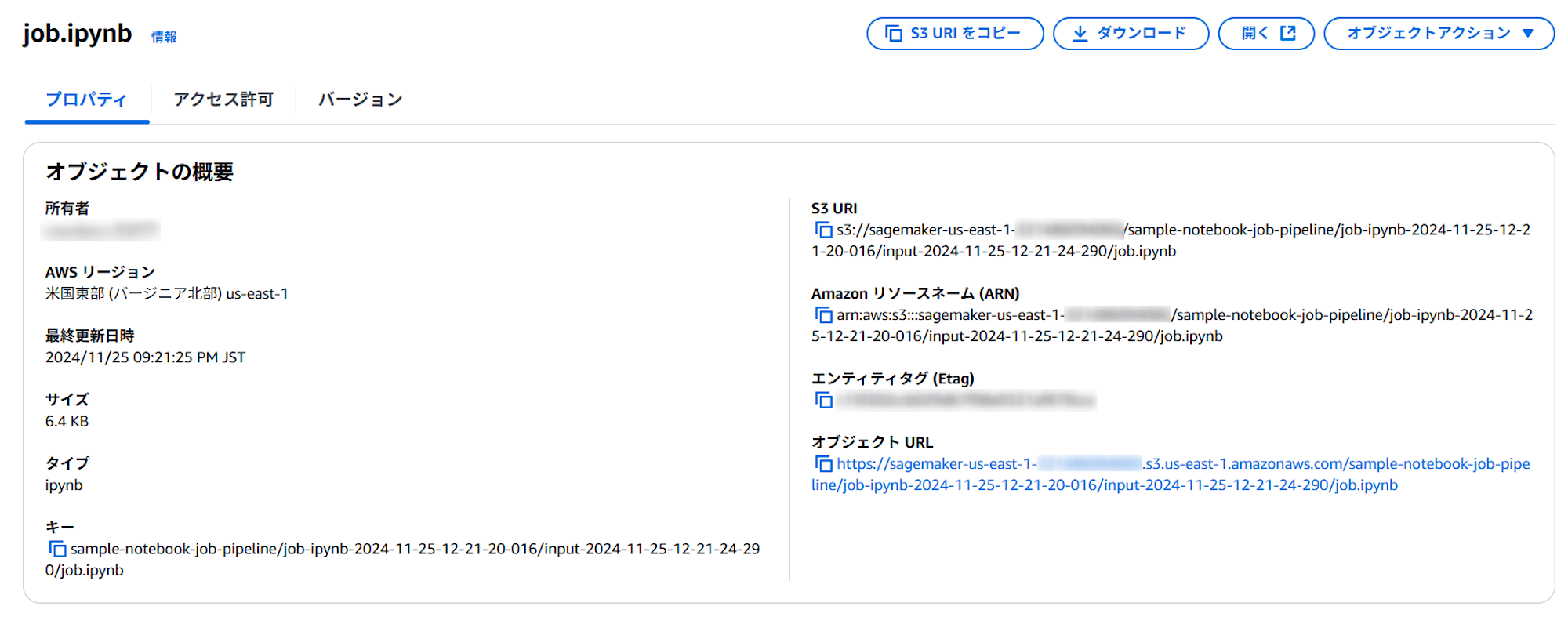
inputDataConfigには以下が設定されており、
"inputDataConfig": [
{
"channelName": "sagemaker_headless_execution_pipelinestep",
"dataSource": {
"s3DataSource": {
"s3DataType": "S3Prefix",
"s3Uri": "s3://{デフォルトバケット}/sample-notebook-job-pipeline/job-ipynb-2024-11-25-12-21-20-016/input-2024-11-25-12-21-24-290",
"s3DataDistributionType": "FullyReplicated"
}
},
"enableFFM": false
}
],
ここには、実行対象となるjob.ipynbが格納されています。
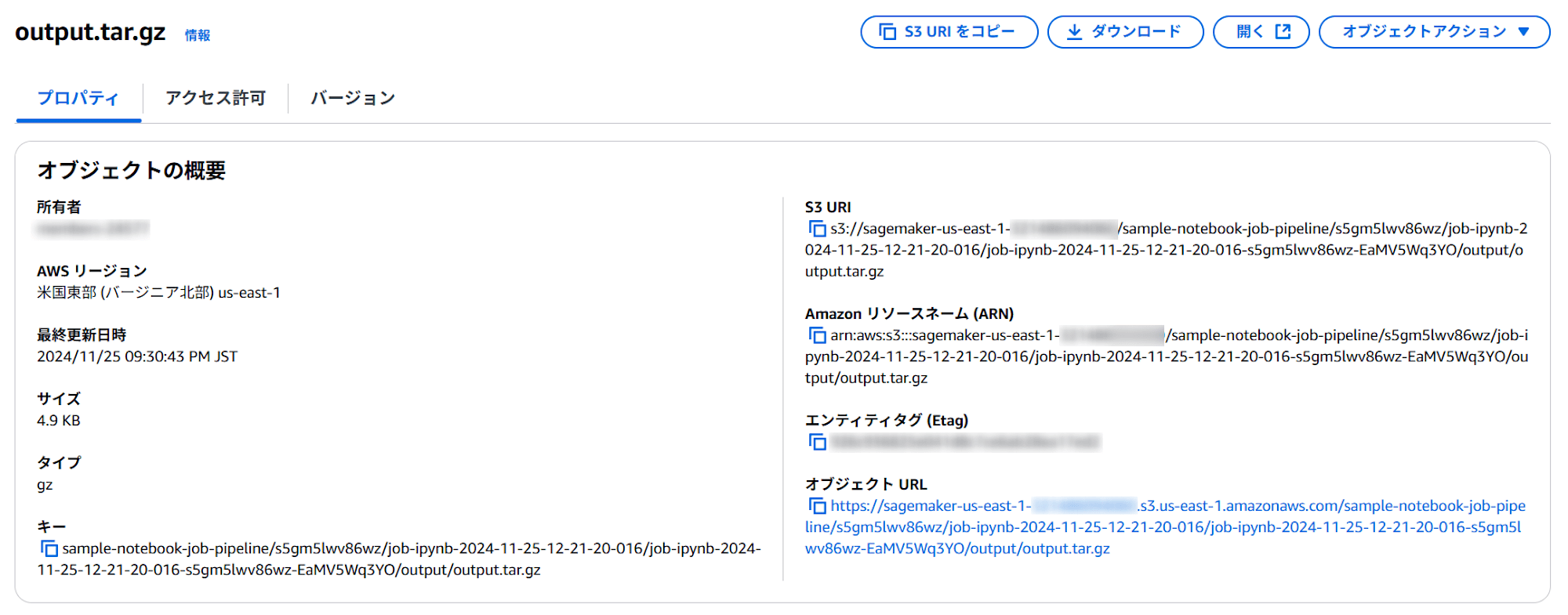
またoutputDataConfigには以下が設定されており、
"outputDataConfig": {
"s3OutputPath": "s3://{デフォルトバケット}/sample-notebook-job-pipeline/s5gm5lwv86wz/job-ipynb-2024-11-25-12-21-20-016",
"removeJobNameFromS3OutputPath": false,
"disableModelUpload": false
},
ここには、出力が圧縮ファイルで格納されています。
この圧縮ファイルを解凍すると以下が格納されています。
job-ipynb-2024-11-25-12-21-20-016.ipynb
sagemaker_job_execution.log
job.ipynb
job.ipynb はアップロードしたノートブックそのものですが、 job-ipynb-2024-11-25-12-21-20-016.ipynb は実行済みのノートブックとなっており、出力セルが記録されています。
また、 sagemaker_job_execution.log は以下のようになっていました。Papermillが使われているようです。
+ TRAINING_OUTPUT_PATH=/opt/ml/output/data
+ TRAINING_FAILURE_PATH=/opt/ml/output/failure
+ SM_EXECUTION_INPUT_PATH=/opt/ml/input/data/sagemaker_headless_execution_pipelinestep
+ SM_EXECUTION_SYSTEM_PATH=/opt/ml/input/data/sagemaker_headless_execution_system
+ export SM_PAPERMILL_INPUT=/opt/ml/input/data/sagemaker_headless_execution_pipelinestep/job.ipynb
+ SM_PAPERMILL_INPUT=/opt/ml/input/data/sagemaker_headless_execution_pipelinestep/job.ipynb
+ mkdir -p /opt/ml/output/data
+ SM_EXEC_STEP='Prepare the input notebook'
+ '[' -f /opt/ml/input/data/sagemaker_headless_execution_pipelinestep/job.ipynb ']'
+ cp /opt/ml/input/data/sagemaker_headless_execution_pipelinestep/job.ipynb /opt/ml/output/data
+ '[' 1.0 '!=' 1.0 ']'
+ SM_EXEC_STEP='detecting python runtime'
+ '[' '!' -z '' ']'
++ which python
+ PYTHON_EXECUTABLE=/opt/conda/bin/python
+ echo 'Python executable: /opt/conda/bin/python'
Python executable: /opt/conda/bin/python
++ /opt/conda/bin/python -c 'import sys; version=sys.version_info[:3]; print("{0}.{1}".format(*version))'
+ DEFAULT_PYTHON_VERSION=3.11
+ '[' true = true ']'
+ echo 'EFS Simulation is skipped.'
EFS Simulation is skipped.
++ id -u
+ [[ 1000 -eq 0 ]]
+ sparkmagic_config_file=/etc/sparkmagic/config.json
+ '[' -e /etc/sparkmagic/config.json ']'
+ SM_EXEC_STEP='running LCC script as init-script'
+ cd /opt/ml/input/data/sagemaker_headless_execution_pipelinestep
+ '[' '!' -z '' ']'
+ echo 'No lcc-init-script is specified.'
No lcc-init-script is specified.
+ SM_EXEC_STEP='running init script'
+ '[' '!' -z '' ']'
+ echo 'No init-script is specified.'
No init-script is specified.
+ SM_EXEC_STEP='executing notebook'
+ export SM_PAPERMILL_PARAMS_PATH=/opt/ml/input/config/hyperparameters.json
+ SM_PAPERMILL_PARAMS_PATH=/opt/ml/input/config/hyperparameters.json
+ export SM_PAPERMILL_OUTPUT=/opt/ml/output/data/job-ipynb-2024-11-25-12-21-20-016.ipynb
+ SM_PAPERMILL_OUTPUT=/opt/ml/output/data/job-ipynb-2024-11-25-12-21-20-016.ipynb
+ export SM_PAPERMILL_FAILURE_FILE=/opt/ml/output/failure
+ SM_PAPERMILL_FAILURE_FILE=/opt/ml/output/failure
++ echo /opt/ml/output/data/job-ipynb-2024-11-25-12-21-20-016.ipynb
++ sed 's|\.|-|g; s|:|-|g; s|-*ipynb$|.ipynb|g;'
+ export SM_PAPERMILL_OUTPUT=/opt/ml/output/data/job-ipynb-2024-11-25-12-21-20-016.ipynb
+ SM_PAPERMILL_OUTPUT=/opt/ml/output/data/job-ipynb-2024-11-25-12-21-20-016.ipynb
+ papermill_process=28
+ wait 28
+ /opt/conda/bin/python -m sagemaker_headless_execution_driver.notebookrunner
Use Kernel python3 to execute job.ipynb with output to /opt/ml/output/data/job-ipynb-2024-11-25-12-21-20-016.ipynb. Params: {}
Executing: 0%| | 0/4 [00:00<?, ?cell/s]
Executing: 25%|██▌ | 1/4 [00:00<00:02, 1.09cell/s]
Executing: 50%|█████ | 2/4 [01:06<01:18, 39.23s/cell]mnist_cnn.pth
Executing: 100%|██████████| 4/4 [01:08<00:00, 15.25s/cell]
Executing: 100%|██████████| 4/4 [01:10<00:00, 17.56s/cell]
Execution complete
Output notebook was generated
+ echo 'Papermill process complete, saving notebook output in the following formats: '
Papermill process complete, saving notebook output in the following formats:
+ save_papermill_output_as_formats
+ '[' -f /opt/ml/output/data/job-ipynb-2024-11-25-12-21-20-016.ipynb ']'
+ export_formats=("$@")
+ echo 'Notebook execution is complete'
Notebook execution is complete
+ exit_code=0
+ exit_hook 0
+ set +x
まとめ
いかがでしたでしょうか。
今回はNotebook Job単体のpipelinesで検証しましたが、前処理やモデル登録などと組み合わせて学習・推論パイプラインにも組み込むことが可能です。
今後そちらの紹介もしていきたいと思います。
本記事がご参考になれば幸いです。









